GENES BIOSINTÉTICOS


G Adithi y M Y Sreenivasa*
Laboratorio de Micología Aplicada, Departamento de Estudios en Microbiología, Universidad de Mysore, Mysuru, India
*Autor correspondiente: sreenivasamy@gmail.com; mys@microbiology.uni-mysore.ac.in
Los hongos micotoxigénicos y sus micotoxinas son una de las principales causas de contaminación en cereales, alimentos, piensos para animales y aves.
Las principales especies de hongos incluyen Aspergillus, Fusarium y Penicillium
Los metabolitos secundarios secretados por estos hongos toxigénicos son micotoxinas, que tienen un grave impacto en la salud humana y animal, causando carcinogenicidad, genotoxicidad, nefrotoxicidad, hepatotoxicidad y trastornos reproductivos.
Las principales micotoxinas son las aflatoxinas, las fumonisinas, los tricotecenos, la zearalenona y las ocratoxinas, que son químicamente estables y no se degradan fácilmente mediante el procesamiento normal o mediante autoclave, por lo que terminan en la cadena alimentaria (Adithi et al., 2022).
Los componentes genéticos de los hongos siempre desempeñan un papel fundamental en la biosíntesis de metabolitos secundarios, incluidas las micotoxinas.


Comprender los genes biosintéticos de las micotoxinas y su regulación se ha convertido en algo fundamental para dilucidar su expresión y control. Esta información puede ser útil para desarrollar enfoques eficaces para la descontaminación de micotoxinas en alimentos y piensos.
La identificación y manipulación de genes biosintéticos y vías reguladoras clave ayudan a desarrollar métodos de control innovadores y específicos para inhibir la biosíntesis de estas toxinas en las plantas y sus productos, garantizando la seguridad alimentaria (Deepa et al., 2016).
Características generales de los clústeres de genes biosintéticos de micotoxinas
En los hongos, los grupos de genes biosintéticos (BGCs) son grupos de genes que controlan la producción de micotoxinas, lo que confirma que la vía funciona de manera coordinada, mientras que la poliquetida sintasa (PKS) actúa como gen principal para la mayoría de las micotoxinas (Proctor et al., 2008; Caceres et al., 2020; Liew et al., 2023)
Estos grupos también incluyen reguladores específicos de vías y múltiples factores de transcripción.
Contienen transportadores responsables de exportar toxinas fuera de la célula para reducir la autotoxicidad.
Los BGCs también son muy flexibles, ya que presentan pérdidas o reordenamientos genéticos que explican las variaciones en la producción de toxinas entre especies y cepas (Sultana et al., 2019).
Genes biosintéticos por tipos de micotoxinas
En las especies de Aspergillus, la producción de aflatoxinas está controlada por dos genes clave, aflR y aflS, que codifican un factor de transcripción con dedos de zinc que activa los genes de la vía de señalización y un coactivador que potencia la función de aflR, respectivamente (Meyers et al., 1998).
Hallazgos recientes muestran que aflS, además de su función como coactivador, también ayuda a estabilizar las interacciones entre AflR y el ADN, y que su pérdida puede afectar a la producción de aflatoxinas (Caceres et al., 2020).

La relación entre aflS y aflR varía en respuesta a factores ambientales como la actividad del agua y la temperatura, lo que contribuye a cambios en los niveles de toxinas y los tipos de aflatoxinas producidas (Medina et al., 2017) (Figura 1).
Figura 1. Representación del grupo biosintético de aflatoxinas en Aspergillus flavus. Las flechas indican la dirección de la transcripción génica. Las flechas grises representan genes reguladores, mientras que las flechas azules indican genes estructurales (Serna et al., 2020).

GENES CLAVE RESPONSABLES DE LAS FUMONISINAS (GENES FUM 1, 6, 8
Y 21)
Las fumonisinas se encuentran entre las principales micotoxinas secretadas por las especies de Fusarium, y son contaminantes comunes de la mayoría de los cereales y granos (Dass et al., 2007; Sreenivasa et al., 2011).
El grupo de genes FUM regula la biosíntesis de estas toxinas, siendo FUM1, FUM6, FUM8 y FUM21 los genes clave (Glenn et al., 2008) (Figura 2).
FUM1 codifica una poliquetida sintasa responsable del ensamblaje de la cadena principal.
FUM6 y FUM8 median la hidroxilación y la metilación, ambas esenciales para la actividad de la toxina.
FUM21 actúa como factor regulador de la transcripción.

La alteración de estos genes reduce significativamente o elimina la producción de fumonisina.
Figura 2. Representación del grupo de genes biosintéticos de la fumonisina en Fusarium verticillioides. Las flechas indican la posición y la orientación transcripcional de los genes. La flecha gris representa el gen regulador. En Fusarium proliferatum y Fusarium oxysporum, el orden y la orientación de los genes son similares, aunque las distancias entre ellos pueden variar (Alexander et al., 2009).

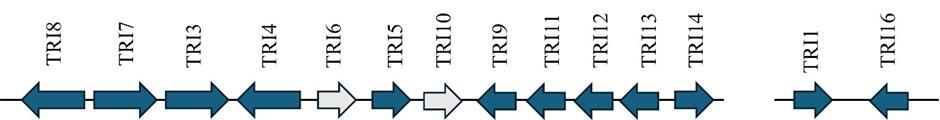
GENES CLAVE RESPONSABLES DE LOS TRICOTECENOS (GRUPOS TRI, TRI5, TRI4, TRI6, TRI7, TRI8, TRI11 Y TRI13)
Los tricotecenos son micotoxinas sesquiterpenoides tóxicas producidas por especies de Fusarium que suponen un grave riesgo de contaminación alimentaria.
La biosíntesis de tricotecenos está controlada por el grupo de genes TRI.
TRI5 codifica la tricodieno sintasa, iniciando la formación de tricotecenos.
TRI4 cataliza múltiples etapas de oxigenación.
TRI6 es un gen regulador que controla la expresión de los grupos.
TRI7, TRI8, TRI11 y TRI13 codifican enzimas que modifican la cadena principal del tricoteceno mediante reacciones como la hidroxilación, la acetilación y la epoxidación, produciendo diversos análogos (Proctor et al., 2008; Caceres et al., 2020)
En conjunto, estos genes coordinan el proceso biosintético y dan lugar a la secreción de tricotecenos de tipo A y tipo B (Figura 3).

Figura 3. Representación del grupo biosintético del tricoteceno y del grupo TRI1-TRI16 en Fusarium, que conduce a la formación de la toxina T-2. Las flechas grises representan genes reguladores, mientras que las demás flechas indican la posición y la orientación transcripcional de los genes (Alexander et al., 2009).

GENES CLAVE RESPONSABLES
(PKS 4, 13 Y ZEB1, ZEB2)
DE LA ZEARALENONA
La zearalenona (ZEN) es una micotoxina estrogénica producida por especies de Fusarium que contamina los cereales y sus productos derivados (Yu et al., 2022)
La biosíntesis de la zearalenona depende de los genes de la poliquetida sintasa (PKS) y de los factores de transcripción reguladores.
PKS4 y PKS13 catalizan el ensamblaje de la cadena principal del policétido a partir de unidades de acetil-CoA, formando el precursor del ZEN (Perincherry et al., 2019).
La expresión de estos genes está regulada por los factores de transcripción clave ZEB1 y ZEB2, que coordinan el metabolismo secundario para una producción eficiente de ZEN (Villani et al., 2019).

La expresión controlada de PKS4, PKS13, ZEB1 y ZEB2 desempeña un papel fundamental en la minimización de la contaminación por ZEN en alimentos, piensos y otros productos agrícolas (Figura 4).
Figura 4. Representación del grupo de genes ZEN que muestra su organización genómica y las regiones flanqueantes en Fusarium graminearum. La dirección de las flechas indica la posición y orientación previstas de la transcripción de cada gen/ORF. El área enmarcada comprende los genes necesarios para la biosíntesis de ZEN en F. graminearum (Nahle et al., 2021).

PKS13: Sintasa de policétidos
GENES CLAVE RESPONSABLES DE LA OCRATOXINA A (AcOTApks GENES PKS)
Las especies Aspergillus y Penicillium secretan ocratoxina A (OTA), responsable de la contaminación de cereales y otros productos agrícolas.
La biosíntesis de la ocratoxina implica una serie compleja y coordinada de procesos enzimáticos en los que la poliquetida sintasa, AcOTApks, desempeña un papel fundamental (Wang et al., 2016).
Este gen codifica una enzima PKS responsable de iniciar la condensación de unidades de acetato y malonato, lo que da como resultado el núcleo de dihidroisocumarina de la OTA (Gallo et al., 2012). El proceso se consolida aún más con la incorporación de L-fenilalanina y la posterior cloración para completar la molécula de OTA (El Khoury y Atoui, 2010)
La biosíntesis de OTA también está regulada por sintasas de péptidos no ribosomales y enzimas clorantes, aunque aún no se ha dilucidado completamente el mecanismo.
Los hallazgos actuales proporcionan información sobre la compleja red genética y enzimática necesaria para la producción de OTA (Figura 5)

Figura 5. Representación del grupo biosintético de la ocratoxina en Aspergillus carbonarius. La flecha gris representa el gen regulador, mientras que las demás flechas indican la dirección de la transcripción génica (Serna et al., 2020).

hal: Halogenasa; bZIP: región básica/cremallera de leucina (factor de transcripción); p450: monooxigenasa citocromo p450; NRPS: sintetasa de péptidos no ribosomales; PKS: sintasa de policétidos





